

科研动态

近日,上海交通大学樊春海、宋萍、左小磊团队在Nature Communications期刊上发表题为“Massively parallel homogeneous amplification of chip-scale DNA for DNA information storage (MPHAC-DIS)”的最新研究。该研究开发了基于热力学自由能驱动的高通量引物设计方法(MPHAC),实现了低浓度核酸序列的平行、均匀扩增。基于该方法构建的DNA存储系统与文本、图片、视频等多模态文件类型兼容,并以99%上解码正确率对任意文件子集进行随机访问。
随着全球数据量的指数级增长,DNA以其超高存储密度、长半衰期以及低能耗优势,成为解决大规模数据存储需求的重要突破口。然而,当前DNA信息存储仍面临显著挑战,包括高昂的DNA合成与测序成本等问题。基于芯片的大规模DNA合成技术可同时合成数百万条DNA分子,极大降低了合成成本,但其产物浓度通常仅为飞摩尔级,远低于二代测序所需的纳摩尔级浓度。传统扩增方法因效率差异,容易产生扩增偏倚,影响数据均一性和测序深度,导致数据读出的准确性下降及测序成本升高。
针对上述问题,团队开发了一种基于热力学控制的均一扩增方法(MPHAC)。该方法通过严格的热力学能控制,辅以GC含量筛选、二级结构检查、引物二聚体过滤、BLAST等手段,实现了对目标序列的高灵敏、高均一性扩增(图1)。相比传统定长引物设计,根据模拟结果,该方法拥有更均匀的扩增效率(Fold-80由3.2降至1.0),在大规模随机访问时最高可使成本下降4个数量级。基于该方法构建的DNA存储系统与多种文件类型兼容,并实现99%以上解码正确率。在此基础上,进一步结合对抗神经网络用于数据修复,最低可在1×测序深度下实现80%正确率。
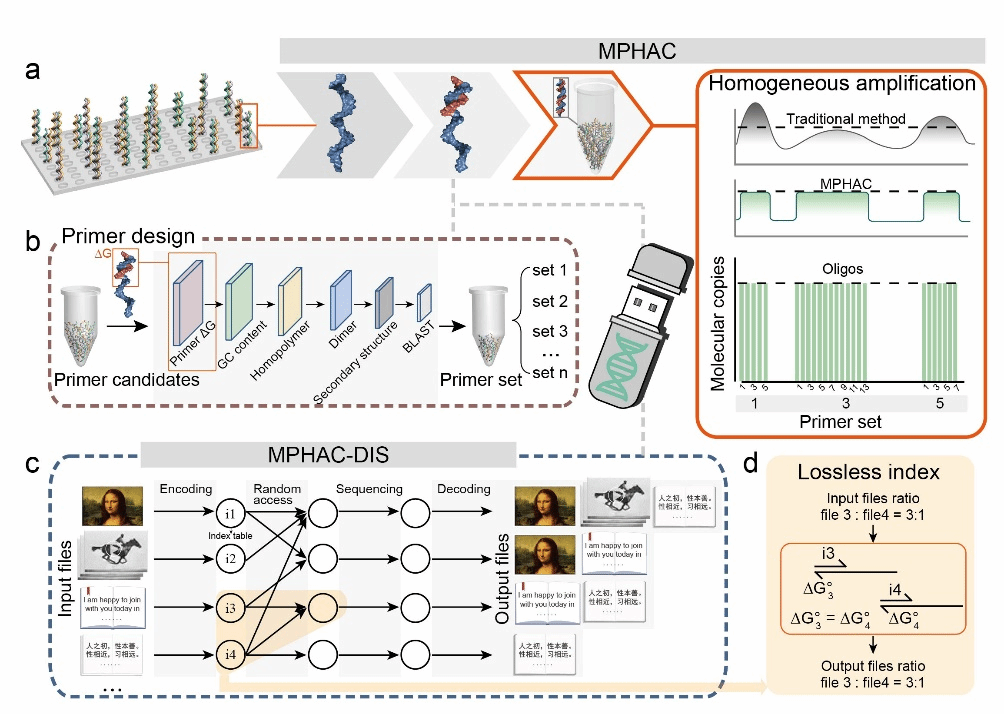
图1 MPHAC与MPHAC-DIS总览图
团队使用编码自校正查找表的方式,将ASCII文件中每一个字节编码成5个碱基的核酸。通过合成35,406条DNA序列,编码了包含三字经、交大庙门、蒙娜丽莎、登月在内的文本、图片、视频文件,并实现了高效访问与解码(图2)。基于MPHAC优秀的扩增均一性与并行性,其有望推动DNA数据存储的文件存储数目与访问通量迈向新阶段。
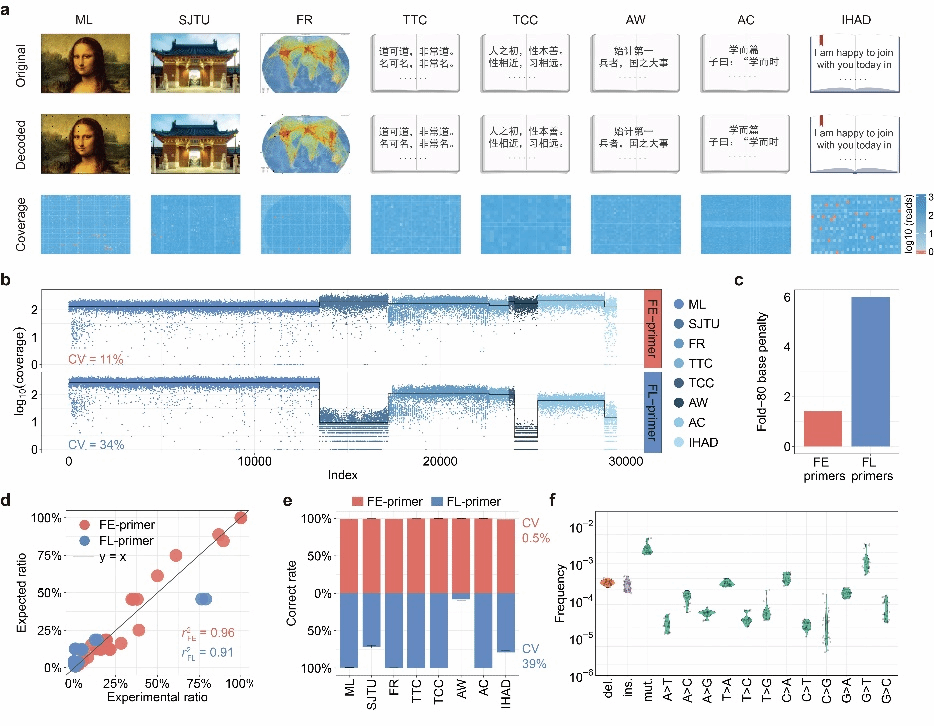
图2 MPHAC-DIS 的均一扩增与无损索引
上海交通大学生物医学工程学院博士研究生翁智与博士后李江雪是该论文的第一作者,化学化工学院/张江高等研究院DNA存储研究中心樊春海院士、生物医学工程学院/张江高等研究院DNA存储研究中心宋萍副教授和仁济医院分子研究院/张江高等研究院DNA存储研究中心的左小磊教授为通讯作者。
这项工作得到了国家重点研发计划、国家自然科学基金、上海市教育委员会“青年领军人才培养计划”项目、上海市科技创新行动计划等的资助。





 交大主页
交大主页